Reação em cadeia da polimerase

A reação em cadeia da polimerase - RCP[1] em inglês polymerase chain reaction - PCR[2] é uma técnica utilizada na biologia molecular para amplificar uma única cópia ou algumas cópias de um segmento de DNA em várias ordens de grandeza, gerando milhares a milhões de cópias de uma determinada sequência de DNA.
Desenvolvida em 1983 por Kary Mullis, que era um funcionário da Cetus Corporation, e também o vencedor do Prêmio Nobel de Química em 1993 junto com Michael Smith,[3] é uma maneira fácil, barata e confiável de replicar repetidamente um segmento específico de DNA, um conceito aplicável a vários campos da biologia moderna e das ciências relacionadas. O PCR é provavelmente a técnica mais utilizada na biologia molecular. Esta técnica é utilizada na pesquisa biomédica, nas técnicas forenses e na arqueologia molecular.
O PCR é agora uma técnica comum e frequentemente indispensável usada em laboratórios clínicos e de pesquisa para uma ampla variedade de aplicações.[carece de fontes] Estas incluem a clonagem de DNA para sequenciação, clonagem e manipulação de genes, mutagênese de genes; construção de filogenias baseadas em DNA, ou análise funcional de genes; diagnóstico e monitoramento de doenças hereditárias; amplificação do DNA antigo;[4] análise de impressões digitais genéticas para o perfil de DNA (por exemplo, na ciência forense e teste de parentesco); e deteção de patógenos em testes de ácido nucleico para o diagnóstico de doenças infecciosas.
A grande maioria dos métodos de PCR dependem do ciclo térmico, o que envolve a exposição dos reagentes a ciclos de aquecimento e resfriamento repetidos, permitindo diferentes reações dependentes da temperatura - especificamente, a fusão do DNA e a replicação de DNA orientada por enzimas - para prosseguir rapidamente várias vezes em sequência. Os iniciadores (fragmentos de DNA curtos) que contêm sequências complementares à região alvo, juntamente com uma polimerase de DNA (por exemplo, Taq polimerase), graças à o qual o método foi designado, permitem amplificação seletiva e repetida. À medida que a PCR progride, o DNA gerado é usado como um modelo para replicação, colocando em movimento uma reação em cadeia na qual o modelo de DNA original é amplificado exponencialmente. A simplicidade do princípio básico subjacente à PCR significa que pode ser amplamente modificada para realizar uma ampla gama de manipulações genéticas. A PCR não é geralmente considerada um método de DNA recombinante, pois não envolve cortar e colar DNA, apenas amplificação de sequências existentes.
Quase todas as aplicações de PCR empregam uma polimerase de DNA estável ao calor, como a polimerase Taq, uma enzima originalmente isolada da bactéria termofílica Thermus aquaticus. Se a DNA polimerase sensível ao calor for utilizada, ela irá desnaturar todos os ciclos no passo de desnaturação. Antes do uso da Taq polimerase, a DNA polimerase tinha que ser adicionada manualmente a cada ciclo, no que era um processo entediante e dispendioso.[5] Esta DNA polimerase monta enzimaticamente uma nova cadeia de DNA a partir de nucleotídeos livres, os blocos de construção de DNA, usando DNA de cadeia simples como molde e oligonucleótidos de DNA (os iniciadores mencionados acima) para iniciar a síntese de DNA.
O PCR, como a tecnologia de DNA recombinante, teve um enorme impacto[1] nos aspectos básicos e diagnósticos da biologia molecular porque pode produzir grandes quantidades de um fragmento de DNA específico a partir de pequenas quantidades de um modelo complexo. As técnicas de DNA recombinante criam clones moleculares ao conferir a uma sequência específica a capacidade de replicar inserindo-a em um vetor e introduzindo o vetor em uma célula hospedeira. O PCR representa uma forma de "clonagem in vitro" que pode gerar, bem como modificar, fragmentos de DNA de comprimento definido e seqüência em uma simples reação automatizada. Além de suas muitas aplicações na pesquisa biológica molecular básica, o PCR promete desempenhar um papel crítico na identificação de seqüências médicamente importantes, bem como um diagnóstico importante na sua detecção.
Princípios
[editar | editar código-fonte]O PCR amplifica uma região específica de uma cadeia de DNA (o alvo de DNA). A maioria dos métodos de PCR amplifica os fragmentos de DNA entre 0,1 e 10 quilos de base (kbp), embora algumas técnicas permitam a amplificação de fragmentos de até 40 kbp de tamanho. A quantidade de produto amplificado é determinada pelos substratos disponíveis na reação, que se tornam limitantes à medida que a reação progride.[6]
Uma configuração básica de PCR requer vários componentes e reagentes,[7] incluindo:
- um modelo de DNA que contém a região alvo de DNA para amplificar
- uma polimerase de DNA, uma enzima que polimeriza novas cadeias de DNA; A polimerase Taq resistente ao calor é especialmente comum,[8] pois é mais provável que permaneça intacto durante o processo de desnaturação de DNA de alta temperatura
- dois iniciadores de DNA que são complementares das extremidades 3 '(três primeiras) de cada uma das cadeias sentido e anti-sentido do alvo de DNA (a polimerase de DNA só pode se ligar e alongar-se a partir de uma região de DNA de cadeia dupla, sem primers lá não é um local de iniciação de cadeia dupla no qual a polimerase pode se ligar); iniciadores específicos que são complementares da região alvo do DNA são selecionados de antemão e são geralmente feitos sob encomenda em um laboratório ou comprados de fornecedores bioquímicos comerciais
- desoxinucleósido trifosfatos ou dNTPs (às vezes denominados "desoxinucleótidos trifosfatos", nucleotídeos contendo grupos trifosfato), os blocos de construção a partir dos quais a DNA polimerase sintetiza uma nova cadeia de DNA
- uma solução tampão que fornece um ambiente químico adequado para uma atividade ótima e estabilidade da DNA polimerase
- catiões bivalentes, tipicamente íons de magnésio (Mg) ou manganês (Mn); Mg2 + é o mais comum, mas Mn2 + pode ser usado para mutagênese de DNA mediada por PCR, uma vez que uma maior concentração de Mn2 + aumenta a taxa de erro durante a síntese de DNA
- catiões monovalentes, tipicamente iões de potássio (K)
A reação é comumente realizada em um volume de 10-200 μl em pequenos tubos de reação (volumes de 0,2-0,5 ml) em um termociclador. O termociclador aquece e esfria os tubos de reação para atingir as temperaturas exigidas em cada passo da reação. Muitos cicladores térmicos modernos utilizam o efeito Peltier, que permite o aquecimento e o resfriamento do bloco que contém os tubos de PCR simplesmente invando a corrente elétrica. Os tubos de reação de paredes finas permitem uma condutividade térmica favorável para permitir o equilíbrio térmico rápido. A maioria dos termostatos têm tampas aquecidas para evitar a condensação no topo do tubo de reação. Os termocicladores mais antigos que não possuem uma tampa aquecida requerem uma camada de óleo em cima da mistura de reação ou uma bola de cera dentro do tubo.
Fases do PCR
[editar | editar código-fonte]Normalmente, a PCR consiste em uma série de 20–40 mudanças de temperatura repetidas, chamadas ciclos, com cada ciclo geralmente consistindo em duas ou três etapas de temperatura discretas (veja a figura abaixo). O ciclismo é muitas vezes precedido por um único passo de temperatura a uma temperatura muito alta (> 90 °C) e seguido por uma retenção no final para a extensão final do produto ou armazenamento breve. As temperaturas utilizadas e o período de tempo aplicado em cada ciclo dependem de uma variedade de parâmetros, incluindo a enzima utilizada para a síntese de DNA, a concentração de íons bivalentes e dNTPs na reação e a temperatura de fusão (Tm) dos primers .[9]
As etapas individuais comuns à maioria dos métodos de PCR são as seguintes:
- Inicialização: este passo é necessário apenas para polimerases de DNA que requerem ativação de calor por PCR de inicialização a quente.[10] Consiste em aquecer a câmara de reação a uma temperatura de 94–96 °C, ou 98 °C, se forem utilizadas polimerases extremamente termostáticas, que é então mantida durante 1-10 minutos.
- Desnaturação: Este passo é o primeiro evento regular dos ciclos e consiste em aquecer a câmara de reação a 94–98 °C por 20 a 30 segundos. Isso faz com que a fusão do DNA, ou a desnaturação, do modelo de DNA de cadeia dupla rompa as ligações de hidrogênio entre bases complementares, produzindo duas moléculas de DNA de cadeia simples.
- Anelamento: no próximo passo, a temperatura de reação é reduzida para 50-65 °C durante 20-40 segundos, permitindo o anelamento dos iniciadores para cada um dos modelos de DNA de cadeia simples. Dois iniciadores diferentes são tipicamente incluídos na mistura de reação: um para cada um dos dois complementos de cadeia simples contendo a região alvo. Os primers são sequências de cadeia simples, mas são muito menores do que o comprimento da região alvo, complementando apenas sequências muito pequenas na extremidade 3 'de cada vertente. É fundamental determinar uma temperatura adequada para o passo de recozimento porque a eficiência e a especificidade são fortemente afetadas pela temperatura de recozimento. Esta temperatura deve ser suficientemente baixa para permitir a hibridação do iniciador à cadeia, mas suficientemente alta para que a hibridação seja específica, isto é, o iniciador deve unir-se apenas a uma parte perfeitamente complementar da corda e em nenhum outro lugar. Se a temperatura for muito baixa, o iniciador pode se ligar de forma imperfeita. Se for muito alto, o iniciador pode não se ligar. Uma temperatura de recozimento típica é de cerca de 3–5 °C abaixo da Tm dos iniciadores utilizados. As ligações estáveis de hidrogênio entre bases complementares são formadas apenas quando a sequência iniciadora se aproxima muito da sequência do modelo. Durante este passo, a polimerase se liga ao híbrido molde-iniciador e começa a formação de DNA.
- Extensão / alongamento: a temperatura neste passo depende da DNA polimerase utilizada; a temperatura de atividade ideal para a polimerase de DNA termoestável da polimerase Taq (Thermus aquaticus) é de aproximadamente 75–80 °C,[11][12] embora uma temperatura de 72 °C seja comumente usado com esta enzima. Neste passo, a DNA polimerase sintetiza uma nova cadeia de DNA complementar à cadeia de matriz de DNA por adição de dNTPs livres a partir da mistura de reação que são complementares ao modelo na direção 5' a 3', condensando o grupo 5'-fosfato dos dNTPs com o grupo 3'-hidroxi no final da cadeia de DNA nascente (alongada). O tempo preciso necessário para o alongamento depende tanto da polimerase de DNA utilizada quanto do comprimento da região alvo de DNA para amplificar. Como regra geral, na sua temperatura ideal, a maioria das polimerases de DNA polimerizam mil bases por minuto. Em condições ideais (isto é, se não houver limitações devido a substratos ou reagentes limitantes), em cada etapa de extensão / alongamento, o número de sequências alvo de DNA é duplicado. Com cada ciclo sucessivo, os fios originais do modelo mais todas as vertentes recém-geradas tornam-se fios de modelo para a próxima rodada de alongamento, levando a amplificação exponencial (geométrica) da região específica do alvo de DNA.
Os processos de desnaturação, anelamento e alongamento constituem um único ciclo. São necessários vários ciclos para amplificar o alvo de DNA para milhões de cópias. A fórmula usada para calcular o número de cópias de DNA formadas após um determinado número de ciclos é 2n, onde n é o número de ciclos. Assim, um conjunto de reação para 30 ciclos resulta em 230, ou 1.073.741.824 cópias da região original do alvo de cadeia dupla.
Alongamento final: Este único passo é opcional, mas é realizado a uma temperatura de 70–74 °C (a faixa de temperatura necessária para a atividade ideal da maioria das polimerases utilizadas na PCR) durante 5–15 minutos após o último ciclo de PCR para garantir que qualquer DNA de cadeia simples restante seja completamente alongado.
Suporte final: o passo final arrefece a câmara de reação a 4–15 °C por tempo indeterminado e pode ser empregado para o armazenamento a curto prazo dos produtos de PCR.


Aplicações
[editar | editar código-fonte]A PCR encontra sua principal aplicação em situações onde a quantidade de DNA disponível é reduzida. Em teoria, é possível amplificar qualquer DNA. Uma das principais aplicações da PCR é na medicina forense, onde pequenas amostras de DNA retiradas da cena de um crime (pedaços de cabelo que contenham bulbo, gotas de sangue ou saliva, pedaços de pelo ou até mesmo a minúscula quantidade de DNA deixada em uma impressão digital) são amplificadas para serem analisadas pelo método de impressão digital genética. A PCR também é rotineiramente utilizada em procedimentos científicos de biologia molecular como amplificação para gerar mutagênese, detecção de mutações ou preparação de fragmentos de DNA para clonagem (inserção em plasmídeo, por exemplo) como também pode ser utilizada para identificação de patógenos que estão presentes em amostras, como por exemplo a identificação de agentes como Candida sp, Chlamydia trachomatis, HPV (Vírus do papiloma humano) e seus genótipos, HIV, Vírus da Hepatite B, etc. A PCR também é utilizada na paleontologia para o sequenciamento gênico de animais pré-históricos. Também é muito utilizada na identificação de micro-organismos, tendo em vista que apenas 1% dos micro-organismos são cultiváveis e podendo ser isolados. A PCR é o primeiro passo para o posterior sequenciamento. Após obter as sequências geradas pelos equipamentos, pode-se consultar bases de dados na internet para tentar localizar suas possíveis origens, sendo bactérias, archeas ou etc.Elizabeth van Pelt-Verkuil, Alex van Belkum, John P. Hays
Procedimentos
[editar | editar código-fonte]
A reação em cadeia da polimerase é um método muito sensível de análise e por isso é realizado com muito cuidado para evitar contaminações que possam inviabilizar ou tornar errôneo o resultado. O processo consiste basicamente em utilizar os mecanismos da replicação in vitro. Os cientistas então simplificaram ao máximo o processo de polimerização das moléculas. A maquinaria para separar as fitas sense e anti-sense são muito complexas na célula, no lugar utiliza-se a mudança de temperatura. Os ciclos são pensados para disponibilizar o sitio alvo para a ligação dos primers, funcionamento da polimerase e início de um novo ciclo.Bruce A. White Em primeiro lugar, deve-se extrair o material genético da célula ou outro material a ser estudado (exemplo: vestígios de crimes) sem danificá-lo. Normalmente o material extraído é o DNA (ADN), mas pode-se trabalhar com o RNA (ARN) em uma RT-PCR que é um desdobramento da PCR e possui outras aplicações.
Depois de extraído o DNA, a este é adicionada uma mistura (também conhecida como pré-mix) que contém os dNTPs (desoxirribonucleotídeos trifosfatos), que são as bases nitrogenadas ligadas com um três fosfato, os primers (também chamados de oligonucleotídeos ou iniciadores) e a enzima Taq-polimerase em uma solução tampão. Toda esta mistura é colocada no termociclador, o qual faz ciclos de temperatura pré-estabelecidos com tempos exatos específicos para cada reação (fragmento a ser amplificado).Ralph Rapley
Na primeira etapa do ciclo a temperatura é elevada de 94 a 96 °C por pouco tempo para que haja a separação da dupla cadeia de DNA (Desnaturação, quebra das pontes de hidrogênio). Na segunda etapa, a temperatura é reduzida entre 50 a 60 °C dependendo da quantidade de citosina (C) e guanina (G) encontrada no primer, para que os primers se anelem (emparelham) com a fita molde de DNA (anelamento). Na última etapa do ciclo a temperatura é elevada a 72 °C para que a enzima possa funcionar sintetizando a nova molécula (extensão), em seguida um novo ciclo é iniciado. Normalmente são realizados de 25 a 40 ciclos para cada reação na qual a taxa de replicação é exponencial
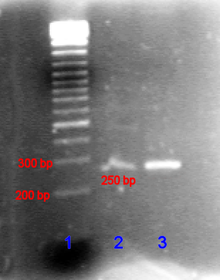
O resultado é analisado através de uma eletroforese em gel de agarose ou de poliacrilamida e depois é interpretado com a ajuda de um profissional competente. Geralmente um padrão de peso molecular é adicionado em uma das fileiras do gel, assim poderá se avaliar o tamanho do fragmento amplificado
Variações da técnica básica de PCR
[editar | editar código-fonte]PCR aninhada
[editar | editar código-fonte]Utilizado para aumentar a quantidade de produto amplificado final ou para aumentar a sensibilidade da técnica. Suas reações requerem dois pares de primers, um par mais externo para a 1a reação e outro interno ao produto da 1a reação. A 1a reação necessita de um maior tempo de extensão devido ao maior tamanho do produto a ser amplificado, em seguida adiciona-se uma alíquota da 1a reação, que servirá de molde na mistura da 1a reação. O produto da 1a reação normalmente não é notado em corrida em gel de agarose. Por outro lado, o produto da 2a reação gerado em grande quantidade o é, por isso pode ser visualizado na corrida eletroforética ou utilizado para sequenciamento.School of Public Health University of California Eva Harris Faculty, Berkeley
Hot Start
[editar | editar código-fonte]A reação de PCR é ativada quando a temperatura atinge 960C. Esse procedimento aumenta a especificidade da PCR, pois a DNA polimerase contém um anticorpo, que se desnatura e ativa a enzima ao atingir esta temperatura. DNA polimerases que não possuem esse inibidor podem amplificar produtos indesejados (inespecíficos) em temperatura ambiente.
Galeria de imagens
[editar | editar código-fonte]-
Clonagem de um gene utilizando-se um plasmídeo.
-
Fingerprint genético: (1)Pai (2)Criança (3)Mãe. A criança somente possui genes (representados por traços pretos) herdados ou da mãe ou do pai.
Ver também
[editar | editar código-fonte]Referências
- ↑ a b Erlich, Henry A. (1 de novembro de 1989). «Polymerase chain reaction». Journal of Clinical Immunology (em inglês). 9 (6): 437–447. ISSN 0271-9142. doi:10.1007/bf00918012
- ↑ Bartlett, John M. S.; Stirling, David (2003). «A Short History of the Polymerase Chain Reaction». Humana Press. Methods in Molecular Biology™ (em inglês): 3–6. ISBN 9780896036420. doi:10.1385/1-59259-384-4:3
- ↑ «Kary B. Mullis - Nobel Lecture: The Polymerase Chain Reaction». www.nobelprize.org. Consultado em 2 de março de 2018
- ↑ J., Ninfa, Alexander (1998). Fundamental laboratory approaches for biochemistry and biotechnology. Bethesda, Md.: Fitzgerald Science Press. ISBN 1891786008. OCLC 38325074
- ↑ Porta, Angela R.; Enners, Edward (1 de abril de 2012). «Determining Annealing Temperatures for Polymerase Chain Reaction». The American Biology Teacher (em inglês). 74 (4): 256–260. ISSN 0002-7685. doi:10.1525/abt.2012.74.4.9
- ↑ Carr, Ana C.; Moore, Sean D. (31 de maio de 2012). «Robust Quantification of Polymerase Chain Reactions Using Global Fitting». PLOS ONE (em inglês). 7 (5): e37640. ISSN 1932-6203. doi:10.1371/journal.pone.0037640
- ↑ Joseph., Sambrook, (2001). Molecular cloning : a laboratory manual 3rd ed. Cold Spring Harbor, N.Y.: Cold Spring Harbor Laboratory Press. ISBN 0879695765. OCLC 45015638
- ↑ Pavlov, Andrey R.; Pavlova, Nadejda V.; Kozyavkin, Sergei A.; Slesarev, Alexei I. «Recent developments in the optimization of thermostable DNA polymerases for efficient applications». Trends in Biotechnology. 22 (5): 253–260. doi:10.1016/j.tibtech.2004.02.011
- ↑ Rychlik, W.; Spencer, W. J.; Rhoads, R. E. (21 de novembro de 1990). «Optimization of the annealing temperature for DNA amplification in vitro ;». Nucleic Acids Research (em inglês). 18 (21): 6409–6412. ISSN 0305-1048. doi:10.1093/nar/18.21.6409
- ↑ Sharkey, David J.; Scalice, Edward R.; Christy, Kenneth G.; Atwood, Susan M.; Daiss, John L. (maio de 1994). «Antibodies as Thermolabile Switches: High Temperature Triggering for the Polymerase Chain Reaction». Bio/Technology (em inglês). 12 (5): 506–509. ISSN 1546-1696. doi:10.1038/nbt0594-506
- ↑ Chien, A.; Edgar, D. B.; Trela, J. M. (setembro de 1976). «Deoxyribonucleic acid polymerase from the extreme thermophile Thermus aquaticus». Journal of Bacteriology. 127 (3): 1550–1557. ISSN 0021-9193. PMID 8432
- ↑ Lawyer, F C; Stoffel, S; Saiki, R K; Chang, S Y; Landre, P A; Abramson, R D; Gelfand, D H. «High-level expression, purification, and enzymatic characterization of full-length Thermus aquaticus DNA polymerase and a truncated form deficient in 5' to 3' exonuclease activity.». Genome Research. 2 (4): 275–287. doi:10.1101/gr.2.4.275
Bibliografia
[editar | editar código-fonte]- ↑ Saiki, R. K., D. H. Gelfand, S. Stoffel, S. J. Scharf, R. Higuchi, G. T. Horn, K. B. Mullis, e H. A. Erlich. Primer-Directed Enzymatic Amplification of DNA with a Thermostable DNA Polymerase. Science 239 (1988): 487-491. PubMed
- ↑ United States Patent 5,656,493 Mullis, et al. August 12, 1997: System for automated performance of the polymerase chain reaction, Patente Número - US 5656493
- ↑ Elizabeth van Pelt-Verkuil, Alex van Belkum, John P. Hays, Principles and Technical Aspects of PCR Amplification , Springer Science & Business Media, 2008 ISBN 1-402-06241-9 (em inglês)
- ↑ School of Public Health University of California Eva Harris Faculty, Berkeley, A Low-Cost Approach to PCR : Appropriate Transfer of Biomolecular Techniques: Appropriate Transfer of Biomolecular Techniques , Oxford University Press, 1998 , ISBN 0-198-02801-6 (em inglês)
- ↑ Bruce A. White, PCR Protocols: Current Methods and Applications, Springer Science & Business Media, 1993, ISBN 1-592-59502-2 (em inglês)
- ↑ Ralph Rapley, PCR Sequencing Protocols, Humana Press, 1996 ISBN 0-896-03344-9 (em inglês)
Ligações externas
[editar | editar código-fonte]- Filme didático, sobre todo o protocolo desta técnica de biologia molecular (1).
- Filme didático, sobre todo o protocolo desta técnica de biologia molecular (2).
- Filme didático, sobre todo o protocolo desta técnica de biologia molecular (3).
- Animação em Flash, Principio da PCR de forma prática e simples de visualizar (1).
- Animação em Flash, Principio da PCR de forma prática e simples de visualizar (2).
- Informação sobre todos os aspectos técnicos dos diferentes tipos de PCR.
- Ciência Forense: exame de ADN - Prof. Emiliano Chemello




