Operon lac
O operon lac (português brasileiro) ou operão lac (português europeu) é um operon requerido para o transporte e o metabolismo da lactose em Escherichia coli e outras bactérias entéricas. O operon lac é regulador por diversos fatores, em particular na disponibilidade de glicose e lactose no meio extracelular. A regulação genética do operon lac foi o primeiro mecanismo complexo de regulação gênica a ser elucidado e é um dos principais exemplos de regulação genética em procariotos. É frequentemente discutido em em aulas introdutórias de biologia molecular e celular por esse motivo. Este sistema de metabolismo da lactose foi usado por François Jacob e Jacques Monod para determinar como uma célula biológica sabe qual enzima sintetizar. Seu trabalho sobre o operon lac lhes rendeu o Prêmio Nobel de Fisiologia em 1965.[1]
Função[editar | editar código-fonte]
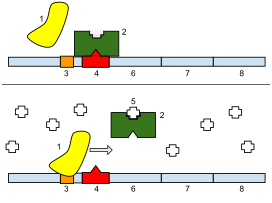
Os operons bacterianos são transcritos policistrônicos que são capazes de produzir várias proteínas a partir de um transcrito de mRNA. O operon lac permite às bactérias uma digestão eficiente da lactose quando a glicose não está disponível. Nesse caso, quando a lactose é necessária como fonte de açúcar para a bactéria, os três genes do operon lac podem ser expressos e suas proteínas subsequentes traduzidas: lacZ, lacY e lacA. O produto do gene lacZ é β-galactosidase que cliva a lactose, um dissacarídeo, em glicose e galactose . lacY codifica beta-galactosídeo permease, uma proteína de membrana que fica embutida na membrana citoplasmática para permitir o transporte celular de lactose para dentro da célula. Finalmente, lacA codifica a galactosídeo acetiltransferase.
Seria ineficiente produzir enzimas quando não há lactose disponível, ou se houvesse no meio uma fonte de energia de mais fácil digestão, como glicose. O operon lac utiliza um mecanismo de controle duplo para assegurar que a célula produza as enzimas somente quando haja lactose no meio.[2] Isso é conseguido através do repressor lac, lacI, que interrompe a produção de enzimas na ausência de lactose,[3] e com a proteína ativadora catabólica (CAP), que auxilia na produção no caso de ausência de glicose no meio. Esse controle duplo faz com que a glicose seja metabolizada pelas bactérias antes que a lactose, o que se conhece pelo nome de diauxia.
Estrutura[editar | editar código-fonte]
O operon lac consiste em três genes estruturais, um promotor, um terminador, um regulador, e um operador.
Os três genes estruturais são lacZ, lacY, e lacA:
- lacZ codifica a β-galactosidase, uma enzima intracelular que degrada o dissacarídeo lactose em glicose e galactose.
- lacY codifica a permease de β-galactosídeos, uma permease de membrana plasmática que bombeia a lactose para dentro da célula através de um gradiente de prótons.
- lacA codifica a transacetilase de β-galactosídeos, uma enzima que transfere um grupo acetil de acetil-CoA a tiogalactosídeos.

Apenas lacZ e lacY parecem ser necessários para o catabolismo da lactose, enquanto que a função do gene lacA é desconhecida. Estes três genes estão organizados como um operon, ou seja, estão dispostos um ao lado do outro na mesma orientação no cromossomo bacteriano e são transcritos como uma única molécula de RNA mensageiro policistrônico.
A transcrição começa quando a RNA polimerase se liga à região promotora, que se localiza a montante (5') dos genes. A partir deste ponto, a polimerase sintetiza uma única molécula de mRNA que inclui a região codificante dos três genes. Cada um dos três genes na fita de mRNA tem sua própria sequência de Shine-Dalgarno, de modo que os genes são traduzidos independentemente.[4] A sequência de DNA do operon lac de E. coli, o mRNA lacZYA e os genes lacI estão disponíveis no GenBank (visualização).
Regulação[editar | editar código-fonte]
O controle para expressão dos genes lac depende da disponibilidade de substrato no meio. Sendo que as proteínas serão produzidas quando ocorre presença de lactose e ausência de glicose. Nesse sentido, para que a célula não tenha gastos energéticos desnecessários, as proteínas reguladoras CAP[5] e lacI são responsáveis por ativar e inativar o operon lac.
Proteína repressora lacI[editar | editar código-fonte]

O gene lacI que codifica o repressor está próximo ao operon lac e é sempre expresso (constitutivo). Esse gene produz a proteína lacI que, na ausência de lactose, se liga fortemente a uma curta sequência de DNA logo a baixo do promotor, próximo ao início do primeiro gene, chamada operador lac. A ligação do repressor ao operador interfere na ligação da RNA polimerase ao promotor e, portanto, o mRNA que codifica LacZ, LacY e LacA é produzido apenas em níveis baixos (basais).
Quando as células são cultivadas na presença de lactose, no entanto, um metabólito da lactose chamado alolactose (produzido através do gene lacZ), liga-se ao repressor, causando um mudança alostérica nessa proteína. Assim, com sua conformação alterada, o repressor é incapaz de se ligar ao operador, permitindo que RNA polimerase transcreva os genes lac, logo, levando a altos níveis de produção das proteínas codificadas.
Esse tipo de regulação é definida como indução negativa, o que quer dizer que o gene é desligado pelo fator regulatório (repressor lacI), ao menos que alguma molécula (lactose) seja adicionada. Uma vez que o repressor é removido, a RNA polimerase então passa a transcrever todos os três genes em mRNA.
Mais recentemente, foi demonstrado que a exclusão do indutor bloqueia a expressão do operon lac quando a glicose está presente. A glicose é transportada para a célula pelo sistema de fosfotransferase dependente de PEP. O grupo fosfato do fosfoenolpiruvato é transferido por meio de uma cascata de fosforilação que consiste nas proteínas PTS (sistema de fosfotransferase) gerais HPr e EIA e nas proteínas PTS específicas da glicose EIIA Glc e EIIB Glc, o domínio citoplasmático do transportador de glicose EII. O transporte de glicose é acompanhado por sua fosforilação por EIIB Glc, levando o grupo fosfato de outras proteínas PTS, incluindo EIIA Glc . A forma não fosforilada de EIIA Glc liga-se à lac permease e evita que ela leve a lactose para a célula. Portanto, se glicose e lactose estiverem presentes, o transporte de glicose bloqueia o transporte do indutor do operon lac.[6]
Proteína ativadora de catabólito (CAP)[editar | editar código-fonte]
O homodímero da proteína ativadora do catabólito (CAP) é o segundo mecanismo de controle do operon lac, que na ausência de glicose auxilia na ligação da RNA polimerase ao promotor. Para que isso aconteça, CAP precisa estar ligada ao monofosfato cíclico de adenosina (cAMP), uma molécula sinalizadora cuja concentração é inversamente proporcional à de glicose. Ou seja, na ausência de glicose, a concentração de cAMP é alta, permitindo o aumento da ligação de CAP-cAMP ao sítio de ligação de CAP (uma sequência de DNA de 16pb, situada a 60pb a montante do sítio de início de transcrição).[7] Dessa forma CAP auxilia a RNA polimerase na ligação ao DNA, aumentando significativamente a produção de β-galactosidase, permitindo que a célula hidrolise a lactose e libere galactose e glicose.
Estrutura do repressor[editar | editar código-fonte]

O repressor lac é uma proteína de quatro porções, um tetrâmero, com subunidades idênticas. Cada subunidade contém um motivo de hélice-volta-hélice (HTH) capaz de se ligar ao DNA. O local do operador onde o repressor se liga é uma sequência de DNA com simetria repetida invertida. Os dois meios-sítios de DNA do operador juntos se ligam a duas das subunidades do repressor. Embora as outras duas subunidades do repressor não apresentem função específica neste modelo, esse funcionamento não foi compreendido por muitos anos.
Eventualmente, foi descoberto que dois operadores adicionais estão envolvidos na regulação lac.[8] Um (O3) fica cerca de -90pb a montante de O1, no final do gene lacI, e o outro (O2) está a cerca de +410 bp a jusante de O1, na parte inicial de lacZ . Esses dois locais não foram encontrados nos primeiros trabalhos porque têm funções redundantes e as mutações individuais não afetam muito a repressão. Mutações únicas para O2 ou O3 têm apenas 2 a 3 efeitos no dobramento. No entanto, sua importância é demonstrada pelo fato de que um duplo mutante defeituoso em O2 e O3 é dramaticamente reprimido.
Mecanismo da indução[editar | editar código-fonte]
O repressor é uma proteína alostérica, ou seja, pode assumir uma de duas conformações ligeiramente diferentes, que estão em equilíbrio entre si. Em uma conformação, o repressor se ligará ao DNA do operador com alta especificidade, e na outra conformação ele perde sua especificidade por essa região. De acordo com o modelo clássico de indução, a ligação do indutor, seja alolactose ou IPTG (um análogo da alolactose), ao repressor afeta a distribuição do repressor entre as duas conformações. Assim, o repressor com indutor ligado é estável na conformação de não ligação ao DNA. No entanto, esse modelo simples apresenta algumas lacunas, já que o repressor está ligado de forma bastante estável ao DNA, mas é liberado rapidamente pela adição do indutor. Portanto, parece claro que um indutor também pode se ligar ao repressor quando este já está ligado ao DNA. Ainda não se sabe exatamente qual é o mecanismo exato dessa ligação.[9]

Análogos da lactose[editar | editar código-fonte]
Têm sido descritos uma gama de derivados ou análogos da lactose que são úteis para trabalhar com o operon lac. Esses compostos são principalmente galactosídeos substituídos, em que a porção glicose da lactose é trocada por outro grupo químico.

- O isopropil-β-D-tiogalactopiranosídeo (IPTG) é frequentemente usado como um indutor do operon lac para experimentos fisiológicos. O IPTG se liga ao repressor e o inativa, mas não é substrato da β-galactosidase. Uma vantagem do IPTG para estudos in vivo é que, uma vez que não pode ser metabolizado por E. coli, sua concentração permanece constante e a taxa de expressão de genes controlados pelo operon lac não é uma variável no experimento. A ingestão de IPTG é dependente da ação da lactose permease em P. fluorescens, mas não em E. coli .[10]

ONPTG 
X-Gal - A fenil-β-D-galactose (fenil-Gal) é um substrato da β-galactosidase, mas não inativa o repressor e, portanto, não é um indutor. Uma vez que as células do tipo selvagem produzem muito pouca β-galactosidase, elas não podem crescer em fenil-Gal como fonte de carbono e energia. Mutantes sem repressor são capazes de crescer em fenil-Gal. Assim, o meio mínimo contendo apenas fenil-Gal como fonte de carbono e energia é seletivo para mutantes repressores e mutantes operadores.
- O tiometil galactosídeo [TMG] é outro análogo da lactose. Estes inibem o repressor lacI. Em baixas concentrações de indutor, ambos TMG e IPTG podem entrar na célula através da permease de lactose. No entanto, em altas concentrações de indutor, ambos os análogos podem entrar na célula independentemente. TMG pode reduzir as taxas de crescimento em altas concentrações extracelulares.[11]

Alolactose - Outros compostos servem como indicadores da atividade da β-galactosidase por mudança de coloração:
- ONPG é clivado para produzir o composto intensamente amarelo, ortonitrofenol e galactose. É comumente usado como um substrato para o ensaio de β-galactosidase in vitro.[12]
- Colônias que produzem β-galactosidase tornam-se azuis por X-gal (5-bromo-4-cloro-3-indolil-β-D-galactosídeo), que é um substrato artificial para B-galactosidase cuja clivagem resulta em galactose e 4-Cl, 3-Br índigo produzindo assim uma cor azul profunda.[13]
- A alolactose é um isômero da lactose e é o indutor do operon lac. A lactose é galactose- (β1-> 4) -glicose, enquanto a alolactose é galactose- (β1-> 6) -glicose. A lactose é convertida em alolactose pela β-galactosidase em uma reação alternativa à hidrolítica. Um experimento fisiológico que demonstra o papel de LacZ na produção do "verdadeiro" indutor em células de E. coli é a observação de que um mutante nulo de lacZ ainda pode produzir LacY permease quando cultivado com IPTG, um análogo não hidrolisável de alolactose, mas não quando cultivado com lactose. A explicação é que o processamento da lactose em alolactose (catalisada pela β-galactosidase) é necessário para produzir o indutor dentro da célula.
Uso na biologia molecular[editar | editar código-fonte]
O gene lac e seus derivados são passíveis de uso como um gene repórter em uma série de técnicas de seleção com base em bactérias, como a análise de dois híbridos, em que o sucesso da ligação de um ativador transcricional a uma sequência promotora específica deve ser determinada.[14] Em placas LB (Caldo Luria-Bertani) contendo X-gal, a mudança de cor de colônias brancas para um tom de azul corresponde a cerca de 20-100 unidades de β-galactosidase, enquanto meios de lactose tetrazólio e de lactose MacConkey têm uma faixa de 100-1000 unidades, sendo mais sensíveis nas porções altas e baixas desta faixa, respectivamente.[14] Como a lactose MacConkey e o meio de lactose tetrazólio dependem dos produtos da degradação da lactose, eles requerem a presença dos genes lacZ e lacY.
Referências[editar | editar código-fonte]
- ↑ Griffiths, Anthony J.F.; Wessler, Susan R.; Carroll, Sean B.; Doebley, John (2015). An Introduction to Genetic Analysis 11 ed. [S.l.]: Freeman, W.H. & Company. pp. 400–412. ISBN 9781464109485
- ↑ McClean, Phillip (1997). «Prokaryotic Gene Expression». ndsu.edu. Consultado em 19 de maio de 2017
- ↑ «Prokaryotic Gene Expression». ndsu.edu. Consultado em 19 de maio de 2017
- ↑ Kennell, David; Riezman, Howard (julho de 1977). «Transcription and translation initiation frequencies of the Escherichia coli lac operon». Journal of Molecular Biology. 114: 1–21. PMID 409848. doi:10.1016/0022-2836(77)90279-0
- ↑ Busby S., Ebright RH. (2001). «Transcription activation by catabolite activator protein (CAP)». J. Mol. Biol. 293: 199–213. PMID 10550204. doi:10.1006/jmbi.1999.3161
- ↑ Görke B, Stülke J (agosto de 2008). «Carbon catabolite repression in bacteria: many ways to make the most out of nutrients». Nature Reviews. Microbiology. 6: 613–24. PMID 18628769. doi:10.1038/nrmicro1932
- ↑ Malan, T. Philip; Kolb, Annie; Buc, Henri; McClure, William (dezembro de 1984). «Mechanism of CRP-cAMP Activation of lac Operon Transcription Initiation Activation of the P1 Promoter». J. Mol. Biol. 180: 881–909. PMID 6098691. doi:10.1016/0022-2836(84)90262-6
- ↑ Oehler, S.; Eismann, E. R.; Krämer, H.; Müller-Hill, B. (1990). «The three operators of the lac operon cooperate in repression». The EMBO Journal. 9: 973–979. PMC 551766
 . PMID 2182324. doi:10.1002/j.1460-2075.1990.tb08199.x
. PMID 2182324. doi:10.1002/j.1460-2075.1990.tb08199.x
- ↑ Griffiths, Anthony JF; Gelbart, William M.; Miller, Jeffrey H.; Lewontin, Richard C. (1999). «Regulation of the Lactose System». Modern Genetic Analysis (em inglês). [S.l.]: W. H. Freeman. ISBN 0-7167-3118-5
- ↑ Hansen LH, Knudsen S, Sørensen SJ (junho de 1998). «The effect of the lacY gene on the induction of IPTG inducible promoters, studied in Escherichia coli and Pseudomonas fluorescens». Curr. Microbiol. 36: 341–7. PMID 9608745. doi:10.1007/s002849900320. Cópia arquivada em 18 de outubro de 2000
- ↑ Marbach A, Bettenbrock K (janeiro de 2012). «lac operon induction in Escherichia coli: Systematic comparison of IPTG and TMG induction and influence of the transacetylase LacA». Journal of Biotechnology. 157: 82–88. PMID 22079752. doi:10.1016/j.jbiotec.2011.10.009
- ↑ «ONPG (β-Galactosidase) test». Setembro de 2000. Consultado em 25 de outubro de 2007. Cópia arquivada em 3 de novembro de 2007
- ↑ Joung J, Ramm E, Pabo C (2000). «A bacterial two-hybrid selection system for studying protein–DNA and protein–protein interactions». Proc Natl Acad Sci USA. 97: 7382–7. PMC 16554
 . PMID 10852947. doi:10.1073/pnas.110149297
. PMID 10852947. doi:10.1073/pnas.110149297
- ↑ a b Joung J, Ramm E, Pabo C (2000). «A bacterial two-hybrid selection system for studying protein–DNA and protein–protein interactions». Proc Natl Acad Sci USA. 97: 7382–7. PMC 16554
 . PMID 10852947. doi:10.1073/pnas.110149297
. PMID 10852947. doi:10.1073/pnas.110149297



