Recombinação V(D)J
Recombinação V(D)J, também conhecida como recombinação somática, é o mecanismo de recombinação genética (do DNA) que ocorre apenas em linfócitos em desenvolvimento durante os primeiros passos da maturação de células B e T. O processo resulta em um repertório diverso de anticorpos (Igs) e receptores de células T (TCRs), encontrados respectivamente em células B e T. O processo é uma característica marcante do sistema imune adaptativo e seu surgimento foi um evento-chave na evolução dos gnatostomados.
A recombinação V(D)J ocorre nos órgãos linfoides primários (medula óssea para células B e timo para células T) e de forma semi-aleatória rearranja os segmentos gênicos V (variable, variável), J (joining, junção) e, em alguns casos, D (diversity, diversidade). O processo resulta em novas sequências de aminoácidos nas regiões de ligação a antígeno de Igs e TCRs que possibilitam o reconhecimento de antígenos de virtualmente todos os patógenos, incluindo bactérias, vírus, parasitas, vermes e até células próprias alteradas, como as presentes no câncer. O reconhecimento pode ser também alérgico (ex: de pólen e outros alérgenos) ou "autorreativo", o que leva a autoimunidade.
Em 1987 Susumu Tonegawa recebeu o Prêmio Nobel de Fisiologia ou Medicina[1] "pela descoberta do princípio genético para geração da diversidade de anticorpos".[2]
Contexto[editar | editar código-fonte]
Os anticorpos humanos ( e os receptores de célula B) são compostos de cadeias pesadas e leves, ambas com regiões constantes (C) e variáveis (V) codificadas por genes em três loci.
| Cadeia | Locus gênico | Domínios |
|---|---|---|
| Cadeia h | Cromossomo 14 | VH, DH, JH |
| Cadeia κ | Cromossomo 2 | VL, JL |
| Cadeia λ | Cromossomo 22 | VL, JL |
Cada gene de cadeia pesada ou leve contem múltiplas cópias de três diferentes tipos de segmentos gênicos para as regiões variáveis dos anticorpos. Por exemplo, a região de cadeia pesada da imunoglobulina contem 2 segmentos constantes (Cμ e Cδ), 44 segmentos "V",[3] 27 segmentos "D" e 6 "J".[4] A cadeia leve possui também 2 constantes ((Cμ and Cδ) e diversos segmentos "V" e "J", mas é desprovida de segmentos "D". Rearranjos de DNA levam a distribuição de apenas 1 cópia de cada tipo de segmento gênico para cada linfócito, gerando assim um repertório enorme de anticorpos, com aproximadamente 3x10<sup)11 possíveis (apesar de algumas serem removidas devido a auto reatividade.
A maioria dos receptores de célula T são compostos de uma cadeia alfa e uma cadeia beta. Os genes do TCR são similares aos das imunoglobulinas, possuindo também múltiplos segmentos V, D e J nas cadeias beta e V e J (mas não D) nas cadeias alfa que são rearranjados durante o desenvolvimento do linfócito, dotando-o de um receptor antigênico único.
A geração de um produto mal-sucedido, que não é capaz de identificar e reagir a estímulos de MHC classe I ou II, leva a célula a apoptose. A autoimunidade é prevenida através da eliminação de linfócitos auto reativos na medula tímica, através de teste com diversos peptídeos peptídeos próprios, produzidos pela ação da proteína AIRE nas células epiteliais reticulares da medula.
Imunoglobulinas[editar | editar código-fonte]
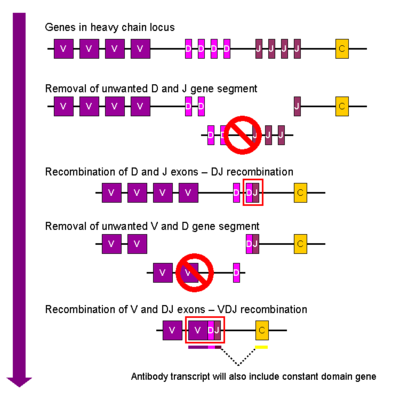
Cadeia Pesada[editar | editar código-fonte]
Na célula B em desenvolvimento, o primeiro evento de recombinação a ocorrer é entre um segmento D e um segmento J do locus da cadeia pesada. Qualquer DNA entre esses dois segmentos é deletado. Essa recombinação D-J é seguida da junção de um segmento V de uma região à montante do complexo DJ, formando um segmento VDJ rearranjado. Todos os outros segmentos entre V e D são também excluídos do genoma. O transcrito primário (RNA pré splicing) é gerado com as regiões VDJ e ambas as cadeias "mu" e "delta" (Cμ and Cδ) (ou seja, o transcrito primário contém os segmentos: V-D-J-Cμ-Cδ). O RNAprimário é então processado, com a adição de uma cauda poli-A após a Cμ e a remoção das bases entre o segmento VDJ e esse segmento constante. A tradução desse mRNA leva a produção da cadeia pesada μ de imunoglobulina.
Cadeia Leve[editar | editar código-fonte]
As cadeias kappa (κ) e lambda (λ) nos loci de cadeia leve rearranjam-se de forma muito semelhante, com a exceção que esses genes não possuem o segmento D. O primeiro passo simplesmente une os segmentos V e J, dando origem a um segmento VJ antes da adição à cadeia constante durante a transcrição primária. A tradução do mRNA processado para cadeia kappa ou lambda resulta na produção da proteína de cadeia leve κ ou λ de imunoglobulina.
A união da cadeia pesada μ com uma das cadeias leves resulta na formação da forma ligada a membrana da imunoglobulina IgM que é expressa na superfície da célula B imatura.
Receptores de célula T[editar | editar código-fonte]
Durante o desenvolvimento dos timócitos, o as cadeias do receptor de célula T (TCR) são submetidas aos mesmo eventos de recombinação ordenada descritos para imunoglobulinas. Recombinação D-J ocorre primeiramente na cadeia beta do TCR. Esse processo pode envolver tanto a junção do segmento Dβ1 a um dos seis segmentos Jβ1 ou do segmento Dβ2 a um dos sete segmentos Jβ2 . A recombinação DJ é seguida, como acima, por rearranjo V - DJ, deleção de todos os segmentos entre os dois grupos e acoplamento ao gene do domínio constante. A transcrição gera, então, a fita de mRNA que permite a tradução da proteína correspondente à subunidade beta do TCR.
O rearranjo da cadeia alfa do TCR ocorre após o da cadeia beta e é semelhante à recombinação V-J descrita para cadeia leve de imunoglobulina. A união das cadeias alfa e beta resulta na produção do TCR αβ, presente na maioria das células T.
Mecanismo[editar | editar código-fonte]
Componentes e enzimas principais[editar | editar código-fonte]
O processo da recombinação V(D)J é mediado pela V(D)J recombinase, com uma diversa coleção de enzimas. As principais enzimas envolvidas são as RAG(Recombination Activation Gene) 1 e 2, a TdT (Terminal deoxynucleotidyl tranferase) e a nuclease Artemis, que faz parte da via de reparo de DNA NHEJ (Non Homologous End Joining).[5] Diversas outras enzimas são conhecidamente envolvidas no processo, como DNA-PK(DNA-dependent protein kinase), XRCC4(X-ray repair cross-complementing protein 4 ), DNA ligase IV, NHEJ1(Non Homologous End Joining factor 1 também conhecida como Cernunos ou fator semelhante a XRCC4) e as DNA polimerases λ e μ.[6] Algumas enzimas envolvidas são específicas de linfócitos (ex: RAG, TdT), enquanto outras são encontradas em outros tipos de células e até de forma ubíqua (ex: componentes da via NHEJ).
Para manter a especificidade de recombinação, a V(D)J recombinase reconhece e se liga a sequências sinal de recombinação (RSSs) que flanqueiam os segmentos V, D e J. As RSSs são compostas de trÊs elementos : um heptâmero de sete nucleotídeos conservados, uma região de espaçamento de 12 a 23 pares de base e um nonâmero de nove nucleotídeos conservados. Enquanto a maioria dos RSSs variam em sequência, as sequências heptaméricas e nonaméricas são CACAGTG e ACAAAAACC respectivamente. Além disso, apesar dos núcleotídeos da região de espaçamento serem variáveis, o tamanaho é relativamente constante..[7][8] O tamanho da região espaçadora corresponde aproximadamente a 1 (12 pbs) ou 2 (23 pbs)voltas da hélice de DNA. Os segmentos a serem recombinados usualmente são ajacentes a espaçadores de tamanho diferente (ex: um possui um 12RSS e o outro um 23RSS).[9] Essa é um passo importante na regulção da recombinação V(D)J.[10]
Processo[editar | editar código-fonte]
A recombinação V(D)J começa quando a recombinase V(D)J (através da atividade da RAG1) se liga a uma RSS que flanqueia um segmento codificante (V, D ou J) e cria um "nick" de fita simples no DNA entre a primeira base da RSS (logo antes do heptâmero) e o segmento codificante. Essa etapa é essencialmente energeticamente neutra ( sem necessidade de hidrólise de ATP) e resulta na formação de grupos 3'-OH e e 5'-fosfato livres na mesma fita. A hidroxila reativa é posicionada pela recombinase para atacar a ligação fosfodiéster da fita oposta produzindo duas terminações de DNA: uma terminação do tipo "hairpin", formando uma volta do lado codificante, e uma terminação de "ponta cega" no lado do segmento sinal. [11] O modelo atual é que o "nick" do DNA e a formação do "hairpin" ocorrem em ambas as fitas simultaneamente, em um complexo chamado de "centro de recombinação"[12][13][14][15]
As terminações de ponta cega são niveladas e ligadas para formar um pedaço circular de DNA contendo todas as sequências entre os segmentos codificantes conhecida como "joint signal" ( apesar da natureza circular, não deve ser confundida com um plasmídeo).
As terminações codificantes são ligadas e posteriormente processadas em variados eventos que, em conjunto, levam a diversidade juncional.[16] A proteína TdT, por exemplo, adiciona núcleotídeos de maneira semialeatória, contribuindo para aumento da diversidade .[17]
Todos esses eventos de processamento levam a uma região de ligação antigênica altamente variável, até mesmo quando os mesmo segmentos gênicos são recombinados. A recombinação permite a geração de imunoglobulinas e receptores de célula T responsivas a antífenos que nem o indivíduo nem seus ancestrais precisam ter necessariamente encontrado, criando espaço para uma resposta imune adaptativa a novos patógenos que se desenvolvem ou que frequentemente se modificam (ex: gripe sazonal) . Contudo, um grande embargo para esse processo é que a sequência de DNA precisa manter seu quadro de leitura em ordem para a proteína final conversar a correta sequência de aminoácidos. Caso a sequência esteja "fora de fase", o desenvolvimento da célula será travado e a célula não conseguirá atingir a maturidade. A recombinação V(D)J é, consequentemente, um processo muito custos e deve ser regulado e controlado de forma rigorosa.
Referências
- ↑ «The Nobel Prize in Physiology or Medicine 1987». nobelprize.org. Consultado em 26 de dezembro de 2014
- ↑ Tonegawa, Susumu (1983). «Somatic Generation of Antibody Diversity». Nature. 309 (5909): 575–581. PMID 6300689
- ↑ Matsuda, F; Ishii, K; Bourvagnet, P; Kuma, K; Hayashida, H; Miyata, T; Honjo, T (1998). «The complete nucleotide sequence of the human immunoglobulin heavy chain variable region locus». The Journal of experimental medicine. 188 (11): 2151–62. PMC 2212390
 . PMID 9841928. doi:10.1084/jem.188.11.2151
. PMID 9841928. doi:10.1084/jem.188.11.2151
- ↑ Li A; Rue M; Zhou J; et al. (junho de 2004). «Utilization of Ig heavy chain variable, diversity, and joining gene segments in children with B-lineage acute lymphoblastic leukemia: implications for the mechanisms of VDJ recombination and for pathogenesis». Blood. 103 (12): 4602–9. PMID 15010366. doi:10.1182/blood-2003-11-3857 Parâmetro desconhecido
|author-separator=ignorado (ajuda) - ↑ Ma, Yunmei; Lu, Haihui; Schwarz, Klaus; Lieber, Michael (setembro de 2005). «Repair of Double-Strand DNA Breaks by the Human Nonhomologous DNA End Joining Pathway: the Iterative Processing Model». Cell Cycle. 4 (9): 1193–1200. PMID 16082219. doi:10.4161/cc.4.9.1977
- ↑ Malu, Shruti; Malshetty, Vidyasagar; Francis, Dailia; Cortes, Patricia (2012). «Role of non-homologous end joining in V(D)J recombination.». Immunologic Research. 54 (1-3): 233–246. PMID 22569912. doi:10.1007/s12026-012-8329-z
- ↑ Ramsden, Dale; Baetz, Kristin; Wu, Gillian (1994). «Conservation of Sequence in Recombination Signal Sequence Spacers». Nucleic Acids Research. 22 (10): 1785–1796. PMID 8208601. doi:10.1093/nar/22.10.1785
- ↑ Cowell, Lindsay; Davila, Marco; Ramsden, Dale; Kelsoe, Garnett (2004). «Computational tools for understanding sequence variability in recombination signals.». Immunological Reviews. 200: 57–69. PMID 15242396. doi:10.1111/j.0105-2896.2004.00171.x
- ↑ van Gent, Dik; Ramsden, Dale; Gellert, Martin (1996). «The RAG1 and RAG2 Proteins Establish the 12/23 Rule in V(D)J Recombination». Cell. 85 (1): 107–13. PMID 8620529. doi:10.1016/s0092-8674(00)81086-7
- ↑ Hiom, Kevin; Gellert, Martin (1998). «Assembly of a 12/23 Paired Signal Complex: a Critical Control Point in V(D)J Recombination». Molecular Cell. 1 (7): 1011–1019. PMID 9651584. doi:10.1016/s1097-2765(00)80101-x
- ↑ Schatz, David; Swanson, Patrick (2011). «V(D)J Recombination: Mechanisms of Initiation». Annual Review of Genetics. 45: 167–202. PMID 21854230. doi:10.1146/annurev-genet-110410-132552
- ↑ Schatz, David; Ji, Yanhong (2011). «Recombination Centres and the Orchestration of V(D)J Recombination». Nature Reviews Immunology. 11 (4): 251–263. PMID 21394103. doi:10.1038/nri2941
- ↑ Curry, John; Geier, Jamie; Schlissel, Mark (2005). «Single-Strand Recombination Signal Sequence Nicks in vivo: Evidence for a Capture Model of Synapsis». Nature Immunology. 6 (12): 1272–1279. PMID 16286921. doi:10.1038/ni1270
- ↑ Agrawal, Alka; Schatz, David (1997). «RAG1 and RAG2 Form a Stable Postcleavage Synaptic Complex with DNA Containing Signal Ends in V(D)J Recombination». Cell. 89 (1): 43–53. PMID 9094713. doi:10.1016/s0092-8674(00)80181-6
- ↑ Fugmann, Sebastian; Lee, AIfred; Shockett, Penny; Villey, Isabelle; Schatz, David (2000). «The RAG Proteins and V(D)J Recombination: Complexes, Ends, and Transposition». Annual Review of Immunology. 18: 495–527. PMID 10837067. doi:10.1146/annurev.immunol.18.1.495
- ↑ Lewis, Susanna (1994). «The Mechanism of V(D)J Joining: Lessons from Molecular, Immunological, and Comparative Analyses». Advances in Immunology. 56: 27–150. PMID 8073949. doi:10.1016/s0065-2776(08)60450-2
- ↑ Benedict, Cindy; Gilfillan, Susan; Thai, To-Ha; Kearney, John (2000). «Terminal Deoxynucleotidyl Transferase and Repertoire Development.». Immunological Reviews. 175: 150–157. PMID 10933600. doi:10.1111/j.1600-065x.2000.imr017518.x
