Ensaio de proteína de Bradford
O ensaio de proteína de Bradford foi desenvolvido por Marion M. Bradford em 1976.[1] É um procedimento analítico espectroscópico rápido e preciso[2] usado para medir a concentração de proteína em uma solução. A reação depende da composição de aminoácidos das proteínas medidas.
Princípio[editar | editar código-fonte]
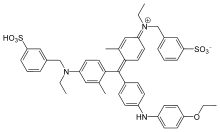

O ensaio de Bradford, um ensaio de proteína colorimétrico, é baseado em uma mudança de absorbância do corante azul brilhante de Coomassie G-250. O corante azul brilhante Coomassie G-250 existe em três formas: aniônico (azul), neutro (verde) e catiônico (vermelho).[3] Sob condições ácidas, a forma vermelha do corante é convertida em sua forma azul, ligando-se à proteína que está sendo testada. Se não houver proteína para se ligar, a solução permanecerá marrom. O corante forma um complexo forte e não covalente com o grupo carboxila da proteína pela força de van der Waals e grupo amino por meio de interações eletrostáticas.[1] Durante a formação deste complexo, a forma vermelha do corante Coomassie primeiro doa seu elétron livre para os grupos ionizáveis na proteína, o que causa uma ruptura do estado nativo da proteína, consequentemente expondo seus bolsões hidrofóbicos. Esses bolsos na estrutura terciária da proteína ligam-se de forma não covalente à região não polar do corante através da primeira interação de ligação (forças de van der Waals) que posicionam os grupos amina positivos em proximidade com a carga negativa do corante. A ligação é reforçada pela segunda interação de ligação entre os dois, a interação iônica. Quando o corante se liga à proteína, causa uma mudança de 465 nm a 595 nm, razão pela qual as leituras de absorbância são feitas em 595 nm.[4]
Ao contrário de outros ensaios de proteínas, o ensaio de proteína de Bradford é menos suscetível à interferência de vários compostos químicos como o sódio, o potássio ou mesmo carboidratos como a sacarose, que podem estar presentes em amostras de proteínas.[2] Uma exceção digna de nota são as concentrações elevadas de detergente. O dodecil sulfato de sódio, um detergente comum, pode ser encontrado em extratos de proteínas porque é usado para lisar células pela ruptura da bicamada lipídica da membrana e para desnaturar proteínas para SDS-PAGE. Enquanto outros detergentes interferem no ensaio em alta concentração, a interferência causada pelo SDS é de dois modos diferentes, e cada um ocorre em uma concentração diferente. Quando as concentrações de SDS estão abaixo da concentração micelar crítica (conhecida como CMC, 0,00333% W/V a 0,0667%) em uma solução de corante Coomassie, o detergente tende a se ligar fortemente à proteína, inibindo os sítios de ligação da proteína para o reagente corante. Isso pode causar subestimativas da concentração de proteína em solução. Quando as concentrações de SDS estão acima da CMC, o detergente se associa fortemente com a forma verde do corante Coomassie, fazendo com que o equilíbrio se desloque, produzindo assim mais da forma azul. Isso causa um aumento na absorbância em 595 nm independente da presença de proteína.
Para medir a absorbância de um composto incolor deve ser realizado um ensaio de Bradford. Alguns compostos incolores, como proteínas, podem ser quantificados em uma densidade óptica de 280 nm devido à presença de anéis aromáticos como triptofano, tirosina e fenilalanina, mas se nenhum desses aminoácidos estiver presente, a absorção não pode ser medida em 280 nm.[5]
Vantagens[editar | editar código-fonte]
Muitas soluções contendo proteínas têm a maior absorção em 280nm no espectrofotômetro, a faixa UV. Isso requer espectrofotômetros capazes de medir na faixa UV, o que muitos não conseguem. Além disso, os máximos de absorção em 280 nm requerem que as proteínas contenham aminoácidos aromáticos como tirosina (Y), fenilalanina (F) e/ou triptofano (W). Nem todas as proteínas contêm esses aminoácidos, fato que distorce as medidas de concentração. Se os ácidos nucleicos estiverem presentes na amostra, eles também absorverão a luz a 280 nm, distorcendo ainda mais os resultados. Ao usar o ensaio de proteína de Bradford, pode-se evitar todas essas complicações simplesmente misturando as amostras de proteína com o corante azul brilhante Coomassie G-250 (reagente de Bradford) e medindo suas absorbâncias a 595 nm, que está na faixa visível.[6]
Este ensaio é um dos ensaios mais rápidos realizados em proteínas.[7] O tempo total necessário para configurar e concluir o ensaio é inferior a 30 minutos.[8] Todo o experimento é feito à temperatura ambiente.
Desvantagens[editar | editar código-fonte]
O ensaio de Bradford é linear em um curto intervalo, tipicamente de 0 µg/mL a 2000 µg/mL, muitas vezes tornando necessárias diluições de uma amostra antes da análise. Ao realizar essas diluições, o erro em uma diluição é agravado em outras diluições, resultando em uma relação linear que nem sempre pode ser precisa.
Referências[editar | editar código-fonte]
- ↑ a b Ninfa, Alexander J; Ballou, David P; Benore, Marilee (2008). Fundamental Laboratory Approaches for Biochemistry and Biotechnology. [S.l.]: Wiley. 113 páginas
- ↑ a b Bradford, Marion (1976). «A Rapid and Sensitive Method for the Quantification of Microgram Quantities of Protein Utilizing the Principle of Protein-Dye Binding» (PDF). Analytical Biochemistry. 72 (1–2): 248–254. PMID 942051. doi:10.1006/abio.1976.9999 – via Google Scholar
- ↑ «Quick Start TM Bradford Protein Assay» (PDF). www.bio-rad.com
- ↑ Bradford, Marion M. (maio de 1976). «A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding». Analytical Biochemistry. 72 (1–2): 248–254. PMID 942051. doi:10.1006/abio.1976.9999
- ↑ P., Ballou, David; Marilee., Benore (2010). Fundamental laboratory approaches for biochemistry and biotechnology. [S.l.]: John Wiley. ISBN 9780470087664. OCLC 420027217
- ↑ Ninfa, Ballou, Benore, Alexander J., David P., Marilee (2010). Fundamental Laboratory Approaches for Biochemistry and Biotechnology. United States of America: John Wiley & Sons, Inc. pp. 110, 113. ISBN 978-0-470-08766-4
- ↑ Okutucu, Burcu; Dınçer, Ayşşe; Habib, Ömer; Zıhnıoglu, Figen (1 de agosto de 2007). «Comparison of five methods for determination of total plasma protein concentration». Journal of Biochemical and Biophysical Methods. 70 (5): 709–711. PMID 17597224. doi:10.1016/j.jbbm.2007.05.009
- ↑ «Protein Assay Technical Handbook» (PDF)
