Usuário:Yleite/LUCA

O último ancestral comum universal (LUCA) é o hipotético último ser vivo a partir do qual todos os seres vivos que vivem actualmente na Terra e fósseis conhecidos descendem. Por isso é ancestral comum mais recente de toda a vida actual e conhecida na Terra. Estima-se que tenha vivido 3,6 a 4,1 mil milhões de anos atrás.[1]
Há mais de um século atrás, Charles Darwin afirmou em sua obra, A Origem das Espécies, que: “... provavelmente todos os seres orgânicos que já viveram nessa terra descendem de alguma forma primordial, na qual a vida deu o seu primeiro sopro”. Essa poderosa ideia de que toda a vida na Terra surgiu a partir de um ancestral comum é amplamente aceita entre os biólogos. Apesar de não ser possível afirmar algo concreto sobre o suposto ancestral comum, intuitivamente assume-se que o mesmo seria uma célula solitária simples, muitas vezes comparada a um procarioto, com pouco ou nenhum metabolismo intermediário. Em princípio, então, uma simples árvore genealógica poderia representar as relações entre todos os organismos viventes e extintos, sendo esse último ancestral comum universal (LUCA) a raiz dessa “árvore da vida”. [1] [2][3] [4]
A noção de origem comum é muito mais motivada hoje em dia desde que estudos bioquímicos, de biologia molecular e genéticos têm demonstrado que muitas características moleculares são universais. [3] Essas evidências da existência do LUCA são chamadas homologias universais, das quais pode-se citar:
- DNA como material genético;
- Cópia de DNA utilizando um modelo e um mecanismo de pareamento de bases;
- Transcrição do DNA em RNA usando uma RNA polimerase com um mecanismo catalítico homólogo;
- Tradução do RNA em proteínas usando o código genético de trincas nucleotídicas;
- Uso de uma mistura de RNAs ribossômicos, RNAs transportadores, e proteínas ribossomais na tradução;
- Uso de ATP como armazenamento energético celular e com envolvimento na síntese de DNA e RNA;
- Fechamento das células em uma membrana plasmática através do qual nutrientes e metabólitos podem transitar. [5]
Conhecer o LUCA permite o delineamento das vias evolutivas que resultaram nos três domínios da vida (Bacteria, Archaea e Eukarya) e marca o ponto de partida para a origem da vida. [6] As questões mais importantes que envolvem o LUCA são se os três domínios da vida formam um grupo monofilético e onde encontra-se a raiz da árvore da vida. [7] Como LUCA compreende um grupo extinto, a sua localização deve ser inferida a partir de relações taxonômicas. [6] A raiz da árvore da vida possui três possíveis localizações, uma em cada ramo que resulta em cada um dos domínios, e um dentro de cada um de qualquer dos domínios.[2]
As variações de traços comuns a Bacteria, Archaea e Eukarya podem facilmente ser explicadas como sendo o resultado de uma separação anterior à divergência do LUCA nesses três grandes domínios. Não há restos paleontológicos para testemunhar a existência do LUCA, mas sua natureza pode ser deduzida através de registros moleculares. [8]
Construção e enraizamento da árvore da vida
[editar | editar código-fonte]Cientistas só foram capazes de iniciar a reconstrução da árvore da vida após os anos 60. Antes desse período, desde a época de Aristóteles, as relações entre os organismos eram estimadas a partir de comparações anatômicas, morfológicas ou ambas. Por esta via, obtinha-se muito pouca informação a respeito dos únicos habitantes da Terra da primeira metade a dois terços da história do planeta, já que eram organismos celulares solitários microscópicos. Com isso, não era possível estimar uma filogenia muito clara para tais microorganismos, deixando os cientistas incertos quanto a sequência na qual algumas das inovações mais radicais na estrutura e função das células ocorreram.
Na metade de 1960, Emile Zuckerkand e Linus Pauling do Instituto de Tecnologia da Califórnia conceberam a ideia de incluir genes e proteínas como base para a construção de árvores genealógicas. Através dessa abordagem, chamada filogenia molecular, pode-se construir árvores filogenéticas através da análise da divergência entre sequências de genes e proteínas isoladas para diferentes organismos. [1]
Como foi demonstrado que o RNA pode atuar como material genético e ser biologicamente catalítico, o estudo sobre a origem e evolução inicial da vida da Terra têm focado no mundo de RNA. A hipótese do mundo de RNA seria de que o RNA é anterior ao advento do DNA e da síntese proteica. [9] Uma das maiores conquistas da cladística molecular foi a comparação evolutiva entre sequências de pequenas subunidades de RNA ribossomal (RNAr), o que possibilitou a construção de uma árvore da vida não enraizada. [8] A árvore universal não enraizada que emergiu a partir do RNAr permitiu um breve clarão, embora indireto, a respeito do nosso último ancestral. [4] Essa molécula é o constituinte chave dos ribossomos, as “fábricas” que constroem as proteínas dentro das células, que tornou-se necessário para a sobrevivência das células ao longo do tempo. [1]
Para inferir as características do LUCA, é necessário o enraizamento da árvore da vida, o que determina a posição do nó ancestral. Através da identificação das características que são exclusivas a cada domínio de vida e determinação de quais caracteres e condições dos mesmos são mais ancestrais, pode-se inferir a composição do LUCA. [5]

Pelo fato de a árvore da vida não possuir um grupo externo a todas as formas de vida existentes, pode-se utilizar genes parálogos (genes homólogos resultantes de uma duplicação gênica) para o seu enraizamento. [7] [5] O método padrão para tal procedimento tem sido a construção dessa árvore a partir de genes que duplicaram antes do surgimento do LUCA. A maior parte das árvores de proteínas presentes em todos os três domínios, fornecem uma árvore na qual Archaea e Eukarya formam um grupo irmão, estando a raiz localizada entre Bacteria e Archaea + Eukarya. Isto sugere que o LUCA era semelhante a uma bactéria. Entretanto, análises diferentes com diferentes sequências, caracteres ou métodos, podem resultar em uma localização diferente da raiz. [2] [7] Uma imagem discreta sobre o LUCA só poderá emergir a partir do momento em que muito mais sequências que representam cada um dos domínios da vida tornarem-se disponíveis.[4]
Incongruências filogenéticas podem ser encontradas em toda a árvore da vida, desde a raiz ao interior das principais ramificações e entre os diversos táxons. Ainda não há uma alternativa consistente à filogenia de RNAr, sendo essa filogenia suportada por um número significativo de genes. Contudo, exceções são suficientemente frequentes e estatisticamente sólidas para que as mesmas não possam ser ignoradas. [4]
Esses resultados indicam que é preciso ter cautela quanto a análises que envolvem um pequeno número de genes, pois não se pode esperar que relações entre os organismos da Terra possam ser determinadas de forma não ambígua desta maneira. [2]
Transferência horizontal e complexidade gênica do LUCA
[editar | editar código-fonte]Archaea e Bacteria compartilham uma grande quantidade de genes metabólicos que não são encontrados em eucariotos. [4] Entretanto, esses dois domínios são geneticamente muito diferentes, o que, de fato, é sustentado pela árvore de RNAr. [1] Se esses dois grupos de “procariotos” fossem resultados de uma divisão filogenética primária e seus genes tivessem sido herdados verticalmente (de geração a geração), seu ancestral comum mais recente deveria possuir todos os genes e suas várias funções que estão presentes nesses domínios. Para isso, esse ancestral necessitaria ser totipotente e uma entidade tão rica e complexa quanto qualquer célula existente atualmente, o que iria contra a ideia dos biólogos de um ancestral simples e com capacidades metabólicas limitadas. Além disso, esse ancestral não seria capaz de explicar a ampla distribuição de diversas funções metabólicas nem a origem primitiva da autotrofia implícita a essa distribuição. Tal ancestral também não seria capaz de explicar a forma de sua evolução, de como surgiu, por exemplo, tanta complexidade em um curto período de tempo e como deu origem tão rapidamente aos ancestrais dos atuais domínios de vida. Tudo isso aparentemente ocorreu por aproximadamente em 1 bilhão de anos, o que é pouco quando comparado com a evolução das linhagens descendentes que já se arrastam por cerca de 3 bilhões de anos. Além disso, estruturas complexas integradas tendem a mudar mais lentamente em relação a estruturas mais simples. Esse suposto ancestral totipotente também possuiria fisiologias associadas que nunca foram observadas em conjunto e, nenhuma linhagem moderna, o que seria exigido caso houvesse herança puramente vertical. [4]
Existem claras evidências de que os genomas das linhagens naturais possuem uma natureza em mosaico, com componentes provavelmente derivados de espécies filogeneticamente diferentes e separadas [8]. Os eucariotos, por exemplo, apresentam genes em organelas que claramente, no geral, não têm a mesma herança dos genes nucleares, sendo provavelmente provenientes da captura de alguns genes de bactérias endossimbióticas. Uma transferência horizontal de genes também é bem observada entre os procariotos [4].
Nos últimos anos, análises de um grande número de genomas celulares completamente sequenciados têm revelado maior discrepância entre as árvores de RNAr. A explicação mais razoável para tal quadro é de que a evolução das células não ocorre apenas por herança vertical de genes, mas a transferência horizontal também tem grande importância no processo. Esse tipo de transferência envolve genes únicos ou regiões gênicas inteiras, que, de acordo com Doolitlle, são passados não apenas entre células estritamente relacionadas, mas também entre espécies diferentes. O grande espectro de recombinações têm sido explicado desde transferência de poucos genes via conjugação, transdução ou transformação, até eventos de fusão celular envolvendo organismos de mesmo ou diferentes domínios. Desta forma, o padrão evolutivo não é tão linear quanto Darwin imaginou. [8] [1]
Se não houvesse transferência lateral de genes, todas as árvores de genes apresentariam a mesma topologia, e os genes ancestrais nas raízes de cada árvore estariam presentes no LUCA, uma única célula ancestral.Entretanto, devido à ocorrência de transferência horizontal, muitas árvores gênicas são incongruentes e nunca seria possível considerar o LUCA como sendo uma única célula. [1] [2]

A possibilidade de ter ocorrido transferência horizontal favorece a hipótese progenota de Woese. Se células rudimentares puderam trocar genes livremente, os três domínios da vida originaram-se a partir de um aglomerado de informações genéticas, o que indicaria que a base da árvore da vida teria múltiplos nós. Com isso, talvez a melhor forma de se representar a evolução das espécies seja na forma de uma rede reticulada, com diversas interconexões entre os ramos, o que sugere que não existe uma simples “Árvore da Vida”.
A transferência lateral de genes também pode gerar confusão ao se inferir as propriedades do LUCA, quando métodos como homologias universais ou reconstrução de estados de caracteres são utilizados. Por exemplo, supondo que RNAr tenha evoluído no ramo bacteriano e posteriormente transferido lateralmente para dos demais domínios, a presença compartilhada dessa molécula em todos os domínios poderia levar a uma impressão errada de que RNAr estava presente no LUCA. Desta forma, para inferir as propriedades do LUCA, deve-se determinar se houve transferência lateral para tais características em análise. [5]
É preciso, contudo, se atentar ao fato de que o grau de dificuldade na reconstrução das características do LUCA e o estudo sobre os primeiros eventos evolutivos, depende, em parte, do tamanho da influência da transferência horizontal e de quais genes foram transferidos. [5] Atualmente, existe um grande debate sobre a extensão da transferência horizontal de genes: alguns autores acreditam que houve pouca transferência, enquanto outros favorecem a ideia de ampla transferência horizontal. [2] De acordo com Delaye et al. (2005), por exemplo, nem todas as sequências são igualmente propensas à tal fenômeno, e as espécies de procariotos não foram afetadas da mesma forma pela transferência horizontal. Segundo ele, a maioria dessas transferências ocorrem entre espécies mais próximas, sendo mais raras entre domínios diferentes. De acordo com essa ideia, se a espécie que carrega as sequências ancestrais não era o próprio ancestral comum, era um parente muito próximo a ele. Caso a transferência horizontal de genes não tenha sido tão forte a ponto de ser a principal força para a distribuição de características homólogas entre os três domínios, LUCA deveria ser um organismo complexo, muito similar a uma bactéria existente atualmente. [8]
O quadro geral que tem emergido é de que organismos, principalmente microorganismos, parecem ter grande facilidade em capturar DNA de fontes exógenas. Entretanto, esse DNA raramente se espalha para o resto da população (provavelmente por produzir poucas vantagens mutacionais). Mesmo quando isso acontece, esse DNA exógeno raramente permanece em uma linhagem por um tempo prolongado, pelo fato de que DNA recentemente capturado é frequentemente usado para adaptações para condições ambientais específicas. Desta forma, uma vez que tais condições ambientais mudam, esse DNA desaparece. Como resultado, há uma tendência para que determinadas porções “nucleares” do genoma não sofram tanta transferência horizontal (como por exemplo, genes housekeeping, como proteínas ribossômicas), e regiões genômicas periféricas sejam mais suscetíveis ao processo. Desta forma, tais porções “nucleares” podem ser utilizadas para gerar árvores filogenéticas, e essa ferramenta também torna válida a existência dos três domínios da vida. [5]
Hipótese progenota
[editar | editar código-fonte]De acordo com Koskela & Anilla (2012), deve-se considerar que pensar em um único ancestral comum é conceitualmente confuso, pois remete a um organismo individual em um pedestal especial. Isto é, como se LUCA fosse o primeiro a se desviar de seus ancestrais, de alguma forma significante, para tornar-se o primeiro ser vivo. [3]
Woese (1998) propôs a hipótese progenota para explicar a natureza do LUCA. Segundo essa teoria, as células que existiram nos primórdios da vida na Terra eram muito diferentes das células modernas, a ponto de não poderem ser consideradas organismos. As propriedade do mecanismo de tradução rudimentar limitou grandemente o potencial natural e evolutivo dessas células, pois não permitia a produção de grande parte, se não todos, os tipos modernos de proteínas. Apenas pequenas proteínas que as células primitivas eram capazes de produzir e usar podiam evoluir. Na era em que viveram (“era progenota”), essas entidades celulares, chamadas progenotas, eram as formas de vida mais avançadas.
Nesse período, também não havia um tipo moderno de mecanismo de reparo de replicação genômica. Isso resulta em altas taxas de mutação, o que requer a existência de pequenos genomas.
A estrutura de tais genomas deveria refletir a dinâmica evolutiva primitiva no geral. Woese (1998) comparou a organização do genoma do progenota com o macronúcleo de alguns ciliados atuais, sendo composto por múltiplas cópias de formas cromossômicas pequenas e lineares (mini-cromossomos). Cada cromossomo estava operacionalmente organizado, de forma que genes estrutural e funcionalmente mais semelhantes estavam agrupados juntos. Esses cromossomos individuais eram “semi-autônomos”, assemelhando-se mais a elementos genéticos modernos que os cromossomos modernos.
Pequenos cromossomos são importantes pois evitam que cada evento de replicação resulte em um grande número de mutações e permitem a existência de mecanismos simples de replicação e transcrição. A multiplicidade cromossômica, por sua vez, gera redundância genética, o que garante a funcionalidade dos genes mesmo caso algum seja silenciado. [4]
Essas células iniciais, cada uma contendo relativamente poucos genes, diferiram de várias maneiras. A comunidade LUCA provavelmente era formada de diversos tipos metabólicos, mas a maiorheterogeneidade estava refletida de forma estrutural, por exemplo, na coexistência de células fagocitárias e não fagocitárias, assim como, talvez, também tipos intermediários. Além da segregação aleatória dos cromossomos (na divisão celular), a organização operacional é selecionada principalmente pela transferência horizontal de genes, e elementos genéticos móveis são adequados para esse tipo de transferência. Herdar apenas uma parte de uma nova via metabólica apresenta pouco ou nenhum benefício, enquanto a troca livre de genes permitia o compartilhamento de talentos entre seus contemporâneos. [4] [1][10]
A divisão celular era simples, por constrição física da célula, gerando duas metades aproximadamente iguais. Durante o processo, os mini-cromossosmos distribuíam-se aleatoriamente entre as células filhas. Isso reduziria a taxa de mutação no sentido de que células que recebessem o melhor balanço de cópias funcionais teriam vantagem seletiva, sendo selecionadas positivamente. [1] Desta forma, apesar de ser uma “comunidade”, essa população não escaparia da seleção natural [10]. Se os erros de replicação pudessem ser diretamente detectados (por exemplo, por mal pareamento), a correção poderia ser feita diretamente pela destruição do mini-cromossomo em questão, por clivagem de nuclease.[4]
Cromossomos primitivos pequenos, com baixa capacidade genética e imprecisão tanto na tradução quanto na replicação sugere rudimentariedade de tal célula em todos os aspectos. [4] Tal comunidade essencialmente dinâmica e instável, ocupava uma ampla faixa de temperatura, e incorporava ou rejeitava inovações constantemente, presumivelmente por algum processo de fusão entre células desprovidas de envoltórios celulares rígidos. [10] Esses subsistemas eram menos complexos e organizados hierarquicamente, estando menos integrados do que as células modernas atuais. [4] Além disso, a temperatura evolutiva dessas células era considerada alta, ou seja, tais subsistemas genéticos aceitavam grande quantidade de mudanças, o que resultava em grande quantidade de variantes. [11]
O Surgimento dos três domínios da vida
[editar | editar código-fonte]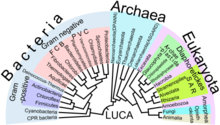
Posteriormente, os subsistemas progenotas aumentaram sua complexidade progressivamente, reduzindo a temperatura evolutiva, enquanto tornavam-se “cristalizados”. De acordo com Kandler (1994, apud Delaye, 2005) as características essenciais para a tradução e o desenvolvimento de vias metabólicas ocorreram antes do primeiro evento de ramificação da árvores da vida, sendo resultado de uma rápida diferenciação dessa comunidade de entidades genéticas. Ao tornarem-se mais complexos, esses subsistemas seriam menos tolerantes a mudanças e assim, ao longo do tempo, estabilizariam em graus diferentes [11][8]. No momento do aparecimento do primeiro Domínio, deve ter existido uma linhagem celular particular que seria um produto das trocas genéticas ocorridas dentro da “comunidade” do LUCA [10]. Eventualmente, essa coleção de células coalesceria nos três domínios básicos conhecidos atualmente, como resultado da diminuição da transferência horizontal de genes e aumento do isolamento genético. [8] [1].
É importante reconhecer que o LUCA não era a única forma de vida em seu período, mas apenas uma espécie ou população dentre diversas outras formas de vida. Essas outras formas seriam uma população de entidades similares a ele que existiram ao longo do mesmo período, entretanto, não deixaram descendentes modernos pois suas linhagens foram extintas. De acordo com Delaye et. al (2005), apesar de tais linhagens não terem sobrevivido, alguns de seus genes permaneceram integrados no genoma do LUCA via transferências horizontal. Segundo esta ideia, o LUCA é um dos últimos resultados evolutivos do tronco de uma árvore de comprimento desconhecido, durante o qual a história de uma longa, mas não necessariamente lenta série de eventos ancestrais, incluindo transferência horizontal de genes, perdas e duplicações gênicas, provavelmente desempenharam um papel significativo no desenvolvimento de genomas complexos. [5] [8]
Inferindo as características do LUCA
[editar | editar código-fonte]Utilizando similaridades nas sequências entre organismos filogeneticamente próximos para validar homologias entre diversas espécies distantes, pode-se atingir sua origem ancestral. Do ponto de vista cladístico, o LUCA seria um inventário de características compartilhadas entre os organismos existentes, localizados no topo dos ramos das árvores moleculares. Para estimar as características do LUCA são usados métodos de Reconstrução do Estado Evolutivo do Caráter, nos quais as características do grupo de organismos de interesse são sobrepostas na árvore evolutiva de tais linhagens. Apesar da dificuldade de se saber de forma confiável os genes que estavam ausentes no LUCA, é bem provável que famílias gênicas representadas em todas as espécies e reinos possuem grande probabilidade de ter estado presente no LUCA. Além disso, domínios que compartilham a mesma linhagem genética possuem grandes chances de compartilhar os mesmos mecanismos e funções moleculares. [11] [5] [8]
Pela comparação entre organismos, pode-se estudar a origem ou a mudança no estado do caráter. Nos métodos mais diretos para a atribuição do estado do caráter no nó ancestral, utiliza-se principalmente o princípio da parcimônia, que visa o menor número de passos evolutivos. [5] Infelizmente, sem saber o conjunto completo de homologias ou as frequências de mutações em cada família gênica, a reconstrução parcimoniosa da constituição gênica do LUCA está sujeita ao erro, especulação e controvérsia. [11]
Em termos de reconstrução do estado do caráter para LUCA, o caso mais simples é aquele em que determinada característica é universal em mesmo estado em todos os organismos. Neste caso, a possibilidade mais direta seria a que todos os organismos, tanto no passado, quanto no presente, teriam a característica. Assim, pode-se inferir que todas as homologias universais estariam presentes no LUCA. [5]
As coisas tornam-se mais complexas quando os traços são universais mas existem em diferentes estados. Por exemplo, a superfamília ATP-loop é representada em 230 ramos ortólogos diferentes, mas apresenta uma grande diversidade de funções. Pode-se inferir que o LUCA teve tal característica, mas também é preciso determinar em que estado este traço estava presente. Saber que este domínio proteico está envolvido na hidrólise de ATP não diz muita coisa à respeito de seu papel funcional no LUCA. Para definir o estado desse caráter, deve-se determinar a polaridade dos caracteres (primitivos ou derivados), o que depende do enraizamento da árvore, pois mostra as relações entre os organismos. [5] [11]
A identificação das características genéticas do LUCA depende de se traçar as relações filogenéticas mais profundas, sendo que estruturas proteicais são mais conservadas através da evolução do que as sequências gênicas. Por exemplo, uma das sequências mais altamente conservadas entre os organismos viventes são as de proteínas que interagem com RNA, sendo que mais de 80% dos domínios conservados correspondem a proteínas com interação direta, como proteínas ribosomais e aminoacil-tRNA sintetases. Entretanto, mesmo com a significativa conservação de mecanismos funcionais e moleculares gerais, alguns domínios de superfamílias ancestrais são altamente diversificados quanto a função, o que dificulta a identificação de suas funções originais. [11] [8]
Em relação à membrana nuclear, acredita-se que a mesma encontrava-se ausente no LUCA. Isto explica-se pelo fato de que seria a explicação mais parcimoniosa na topologia mais amplamente aceita, tendo surgido apenas no ramo que originou os Eukarya e estando ausente tanto nas linhagens de Bacteria quanto Archeae. Desta forma, a membrana é considerada um traço derivado.[5]
Uma análise genômica de 100 espécies identificou 60 genes ubíquos, estando a maioria deles relacionados aos componentes do sistemas de tradução e alguns poucos ao sistema de transcrição, que poderiam confiantemente ser atribuídos ao LUCA. [11]
Possíveis característica do LUCA
[editar | editar código-fonte]A partir desses estudos, dentre algumas das prováveis características presentes no LUCA, pode-se citar:
- Gliceraldeído-3-fosfato desidrogenase e glicerol 3-fosfato quinase: enzimas importantes para a via glicolítica, podendo ter contribuído grandemente para o desenvolvimento do LUCA em ambientes anaeróbicos.
- Transaldolase and transacetolase: enzimas que atuam no ciclo de Calvin e na via das pentoses-fosfato.
- Acetil-CoA: relacionada à síntese de colesterol e de esteroides e síntese e degradação de ácidos graxos.
- α-cetoglutarato desidrogenase e fumarase: participam do ciclo do ácido cítrico.
- Nucleosídeo fosforilase: participa do metabolismo dos nucleotídeos, estando envolvida na via de recuperação de purina e pirimidina.
- Domínios de proteínas estruturais envolvidos em:
- Síntese, reparo, ligação e modificação do DNA;
- Transcrição do DNA;
- Partícula ribossômica;
- Síntese proteica;
- Aminoacil-tRNA sintetase;
- Transferência de grupos metil e NH3 e interconversão de aminoácidos.
- Proteínas com características de chaperonas (envolvidas no reconhecimento entre proteínas, transdução de sinal, regulação gênica,divisão celular, reconhecimento de sinal proteico, e transporte) ou com atividade peptidilpropil isomerase.
- Grupos com atividade relacionadas a biogênese de membrana, transporte de elétrons, e ATP sintase.
- Síntese de DNA, RNA, ou proteínas provavelmente similar ao de bactérias viventes. [11]
- Utilização apenas dos anticódons GNN+ UNN. [6]
- Diversas sequências relacionadas ao metabolismo do açúcar, como genes codificadores de enolases.
- Genes homólogos de tioredoxina (trxB), fosforibosilpirofosfato sintase (prs), and UDP-galactose 4-epimerase (galE), sendo que os genes trxB and prsA estão relacionados a biossíntese de nucleotídeos.
- Várias RNA helicases ATP-dependentes: participação no desenrolamento e rearranjo de moléculas de RNA, incluindo, por exemplo, iniciação da tradução, splicing do RNA, montagem de ribossomos e transporte núcleo-citoplasmático do RNAm. [8]
DNA ou RNA?
[editar | editar código-fonte]Woese (1998) defendeu a ideia de que o LUCA era composto por pequenos fragmentos de RNA. Penny & Poole (1999) afirmam que o passado mais distante que pode ser explorado é o mundo do RNA, onde o RNA tinha tanto a função de codificador quanto de catalisador. A partir disso, ele sugeriu que a tradução foi o primeiro mecanismo a se especializar, pois por ser baseado em RNA, que inclusive precede as proteínas, isso sugere ancestralidade em relação a função. Sua complexidade e universalidade seriam argumentos para uma consolidação precoce. Com isso, as maiores relíquias que podem ser reconstruídas girariam em torno do ribossomo e da tradução. [2] [4]
De acordo com Mushegian and Koonin (1996) (apud Delaye et. al, 2005), a ausência de componentes chave homólogos para a replicação de DNA e biossíntese de nucleotídeos entre Eucarya e Archeae sugere um LUCA era constituído por RNA. [8] Woese afirma que o sistema de replicação genômica não é universalmente distribuído, visto que, por exemplo, não há relação específica ao sistema basicamente comum entre Archaea e eucariotos. Portanto, genomas modernos (relacionados ao DNA) aparentemente surgiram apenas após o estabelecimento dos primeiros domínios, e a evolução para os mecanismos de replicação envolveram inovações importantes que não poderiam se espalhar globalmente. [4]
Por outro lado, Delaye et al. (2005) contesta a ideia de que o DNA evoluiu depois do RNA e das proteínas, pois, isso necessitaria que, como foi sugerido por Woese (1998), a maquinaria de tradução fosse anterior às ribonucleotídeos redutases ou DNA polimerases. No entanto, a grande semelhança das características básicas compartilhados pelas células existentes sugerem que eles devem ter sido integrados no LUCA.
Pelo fato de que todas as células existentes serem constituídas por DNA, Delaye et al (2005) afirma que a hipótese mais parcimoniosa seria a presença desse polímero genético na população do LUCA. Dentre os argumentos para tal possibilidade pode-se citar a presença de similaridades entre sequências compartilhadas por muitos organismos ancestrais. Grandes proteínas encontradas nos três domínios sugerem uma fidelidade considerável no sistema genético operatório do LUCA, mas essa fidelidade é pouco provável de ser encontrado em sistemas genéticos baseados em RNA. [8]
Referências
- ↑ a b c d e f g h i j Doolittle, W. Ford. "Uprooting the tree of life". Scientific American 2000, 282 (6): 90–95.
- ↑ a b c d e f g Penny, D.; Poole, A. “The nature of the Last Universal Common Ancestor”. Current Opinion in Genetics & Development 1999, (9): 672-677.
- ↑ a b c Koskela, M; Anilla, A. “Looking for the Last Universal Common Ancestor (LUCA)”. Genes 2012, (3): 81-87;
- ↑ a b c d e f g h i j k l m n Woese, C. “The Universal Ancestor”. Proceedings of the National Academy of Sciences 1998, 95 (12): 6854 – 6859.
- ↑ a b c d e f g h i j k l Barton, N. H.; Briggs, D. E.G.; Eisen, J. A.; Goldstein, D. B., Patel, N. H. “Evolution”. 1ª edição. Nova Iorque: Cold Spring Harbor Laboratory Press. 2007. Cap. 5. 109-136.
- ↑ a b c Wong, J. T.; Chen, J.; Mat, W.; Ng, S.; Xue, H. “Polyphasic evidence delineating the root of life and roots of biological domains”. Gene 2007, 403 (1-2): 39-52.
- ↑ a b c Cejchan, P.A. “LUCA, or just a conserved Archaeon?: Comments on Xue et al.” Gene 2004, 333 47–50.
- ↑ a b c d e f g h i j k l m Delaye, L.; Becerra, A.; Lazcano, A. “The last common ancestor: what's in a name?” Origins of Life and Evolution of Biospheres 2005, 35 537–54.
- ↑ Hoeppner, M.P.; Gardner, P.P.; Poole, A.M. “Comparative analysis of RNA families reveals distinct repertoires for each domain of life”. PLoS Computational Biology 2012, 8 (11): 1-8
- ↑ a b c d Glansdorff, N.; Xu, Y.; Labedan, B. “The Last Universal Common Ancestor: emergence, constitution and genetic legacy of an elusive forerunner”. Biology Direct 2008, 3 (29): 1-35.
- ↑ a b c d e f g h Ranea, J. A. G.; Sillero, A.; Thornton, J. M.; Orengo, C. A. “Protein Superfamily Evolution and the Last Universal Common Ancestor (LUCA)”. Journal of Molecular Evolution 2006, (63):513–525.
